Компьютерное зрение в микроскопии — различия между версиями
Sashapff (обсуждение | вклад) |
м (rollbackEdits.php mass rollback) |
||
| (не показана 341 промежуточная версия 2 участников) | |||
| Строка 1: | Строка 1: | ||
| − | Компьютерное зрение помогает | + | [[Компьютерное зрение |Компьютерное зрение]] помогает автоматизировать обработку изображений, полученных с помощью микроскопии. С его появлением стало возможным эффективно и с хорошей точностью классифицировать клетки, сегментировать полученные изображения, улучшать их качество и решать другие задачи без участия человека<ref>[https://core.ac.uk/download/pdf/81981016.pdf Gaudenz Danuser {{---}} Computer Vision in Cell Biology, 2011]</ref>. |
| − | |||
= Задачи компьютерного зрения в микроскопии = | = Задачи компьютерного зрения в микроскопии = | ||
| + | На данный момент компьютерное зрение нашло применение в большинстве направлений, где есть необходимость обрабатывать и анализировать изображения. Микроскопия не стала исключением. Теперь задачи, направленные непосредственно на работу с изображениями, можно решить используя методы [[Глубокое обучение|глубокого обучения]], например, построить подходящую [[Сверточные нейронные сети |сверточную нейронную сеть]]<ref>[https://www.researchgate.net/publication/221786123_Computer_Vision_for_Microscopy_Applications Nikita V Orlov {{---}} Computer Vision for Microscopy Applications, 2007]</ref>. | ||
== Классификация клеток == | == Классификация клеток == | ||
| − | [[Файл: | + | [[Файл:Dividing Cell Fluorescence-ru.jpg|left|300px|thumb|Рисунок 1. Пример клетки с различными флуоресцентными маркерами<ref>[https://ru.wikipedia.org/wiki/%D0%A4%D0%BB%D1%83%D0%BE%D1%80%D0%B5%D1%81%D1%86%D0%B5%D0%BD%D1%86%D0%B8%D1%8F_%D0%B2_%D0%B1%D0%B8%D0%BE%D0%BB%D0%BE%D0%B3%D0%B8%D1%87%D0%B5%D1%81%D0%BA%D0%B8%D1%85_%D0%B8%D1%81%D1%81%D0%BB%D0%B5%D0%B4%D0%BE%D0%B2%D0%B0%D0%BD%D0%B8%D1%8F%D1%85 Википедия: Флуоресценция в биологических исследованиях]</ref>.]] |
| − | Классификация клеток является базовой задачей | + | Классификация клеток является базовой задачей микроскопии. Обычно для этого используются изображения, полученные на флуоресцентных микроскопах (рисунок 1), так как классификаторы для изображений с обычных оптических микроскопов не способны отразить биологическое разнообразие различных типов клеток. Клетки можно делить по фазе в клеточном цикле, типу (повержденные или нет, раковые или нормальные), физиологическому состоянию, виду и другим признакам. Для большинства задач классификации уже существуют готовые архитектуры сверточных сетей<ref>[https://uu.diva-portal.org/smash/get/diva2:1334417/FULLTEXT01.pdf Håkan Öhrn {{---}} General image classifier for fluorescence microscopy using transfer learning , 2019]</ref><ref name="phase">[https://www.nature.com/articles/s41467-017-00623-3#supplementary-information Philipp Eulenberg {{---}} Reconstructing cell cycle and disease progression using deep learning, 2017]</ref><ref name="cancer">[https://pubmed.ncbi.nlm.nih.gov/30865716/ Ronald Wihal Oei {{---}} Convolutional neural network for cell classification using microscope images of intracellular actin networks, 2019]</ref><ref name="blood">[https://www.nature.com/articles/s41598-020-59215-9 Ahmed T. Sahlol {{---}} Efficient Classification of White Blood Cell Leukemia with Improved Swarm Optimization of Deep Features, 2020]</ref><ref>[https://www.biorxiv.org/content/10.1101/817544v1.full Samuel Berryman {{---}} Image-based Cell Phenotyping Using Deep Learning, 2019]</ref><ref>[http://www.mva-org.jp/Proceedings/2017USB/papers/06-01.pdf Nan Meng {{---}} Computational single-cell classification using deep learning on bright-field and phase images, 2017]</ref><ref>[https://arxiv.org/pdf/1806.01313.pdf Sachin Mehta {{---}} Y-Net: Joint Segmentation and Classification for Diagnosis of Breast Biopsy Images, 2018]</ref>. |
| + | === Определение фазы клеточного цикла === | ||
| + | Одним из признаков, по которым можно разделить клетки, является определение фазы клеточного цикла, в которой находится клетка. Эта задача имеет практическое применение для обнаружения поврежденных клеток, которые при визуализации будут кластеризоваться отдельно от остальных. Сверточная сеть для решения задачи изображена на рисунке 2. Она обучается на изображениях с флуоресцентными метками, о которых было сказано ранее, и дает на выходе не только классификацию каждой клетки, а также визуализирует процесс клеточного цикла, используя нелинейное уменьшение размерности<ref name="phase"/>. Классификация и визуализация являются всего лишь различными способами интерпретации результатов, поэтому строятся на основе одних и тех же выведенных закономерностей. | ||
| + | [[Файл:Cell cycle classification.png|right|700px|thumb|Рисунок 2. Архитектура сверточной нейронной сети для определения фазы клетки<ref name="phase"/>.]] | ||
| + | Особенностью работы описанной сверточной сети является необходимость разметить только небольшую часть данных, на основании чего она далее учится размечать самостоятельно. Интересно, что при визуализации фазы становятся упорядочены в хронологически правильном порядке, несмотря на то, что информация о порядке фаз не передавалась в сеть напрямую. Это говорит об эффективности использования флуоресцентных меток в качестве категориальных. | ||
| − | + | === Идентификация раковых клеток === | |
| + | [[Файл:microscopy_cnn.png|right|400px|thumb|Рисунок 3. Архитектура сверточной нейронной сети для классификации раковых клеток<ref name="cancer"/>.]] | ||
| + | Другой задачей классификации является обнаружение раковых клеток. Для ее решения используется сверточная нейронная сеть, изображенная на рисунке 3. Она имеет архитектуру VGG-16, в которой дополнительно после каждой функции активации добавлена [[Batch-normalization|пакетная нормализация]] для регуляризации<ref name="cancer"/>. Сеть, как и остальные классификаторы изображений микроскопии, принимает на вход изображения с флуоресцентными метками. | ||
| + | Особенностью при обучении представленной сети является использование из-за недостаточного объема данных [[Глубокое обучение|трансферного обучения]], то есть модель предварительно обучается на другом огромном объеме данных. В данном случае первые 14 слоев сначала обучаются на наборе данных классификации ImageNet, а потом уже происходит обучение для классификации раковых и нормальных клеток. Такая сверточная сеть лучше справляется с задачей классификации клеток по сравнению с экспертом-человеком, особенно на изображениях с недостаточно хорошим качеством. | ||
| − | == | + | === Классификация лейкоцитов === |
| + | [[Файл:Vggnet sessa.jpg|left|400px|thumb|Рисунок 4. Блок-схема подхода к классификации лейкоцитов<ref name="blood"/>.]] | ||
| + | Эта задача отличается от предыдущих тем, что не требует предварительной обработки материалов и использования флуоресцентного микроскопа, для ее решения достаточно изображений с оптического микроскопа. Лейкоциты важно уметь классифицировать, потому что они играют значительную роль в организме человека (иногда в крови могут образоваться злокачественные лейкоциты, которые способны вызывать лейкемию). Автоматическая классификация лейкоцитов может помочь специалистам в лабораториях, где нет достаточного количества квалифицированных сотрудников для проведения анализа крови. | ||
| + | |||
| + | Для решения задачи используется сеть VGGNet, похожая на ту, что идентифицирует раковые клетки, и так же [[Глубокое обучение|трансферное обучение]]. Отличительной деталью является использования для оптимизации статистически усиленного алгоритма сальпового роя (англ. Statistically Enhanced Salp Swarm Algorithm, SESSA)<ref name="blood"/> для выбора наиболее важных функций, извлеченных с помощью сети VGGNet, и удаления сильно коррелированных и шумных функций. Общая структура алгоритма представлена на рисунке 4. Такая оптимизация позволяет сократить время обучения, повысить точность и избавиться от переобучения. Результаты этого гибридного подхода к классификации являются самыми эффективными среди всех известных опубликованных работ по тому же набору данных. | ||
== Сегментация изображений == | == Сегментация изображений == | ||
| + | Задача [[Сегментация изображений|сегментации изображений]], полученных с микроскопа, состоит в том, чтобы аннотировать их, то есть отмечать границы объектов (клеток, ядер). Имея сегментированное изображение, легче проводить дальнейший анализ и изучать конкретные части клетки<ref>[https://www.nature.com/articles/s41524-020-00363-x James P. Horwath {{---}} Understanding important features of deep learning models for segmentation of high-resolution transmission electron microscopy images, 2020]</ref>. | ||
| + | [[Файл:DRAN.jpeg|250px|left|thumb|Рисунок 5. Архитектура сети DRAN для сегментации ядер<ref name="dran">[https://www.frontiersin.org/articles/10.3389/fbioe.2019.00053/full Quoc Dang Vu {{---}} Methods for Segmentation and Classification of Digital Microscopy Tissue Images, 2019]</ref>.]] | ||
| + | === Сеть U-Net === | ||
| + | Для решения задачи сегментации обычно используется модифицированная полносвязная сверточная сеть U-Net<ref>[https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/ Olaf Ronneberger— Reconstructing cell cycle and disease progression using deep learning, 2015]</ref>. Сеть U-Net получила широкое распространение благодаря способности последовательно распознавать как большие, так и мелкие частицы, а также устойчивости к различным условиям визуализации и наборам данных. Также она показывает хорошие результаты, даже если размер набора даных для обучения небольшой, что является частой проблемой анализа изображений, полученных с микроскопа. Однако минусом сети U-Net является ограничение на размер входного изображения, в то время как разрешение микроскопических изображений только растет с течением времени. | ||
| + | |||
| + | === Сегментация ядер клеток === | ||
| + | Сеть U-Net хоть и является универсальной, но для решения некоторых задач удобнее использовать более специализированные сети. Для задачи точной сегментации ядер клеток строится сеть глубокой остаточной агрегации (англ. Deep Residual Aggregation Network, DRAN)<ref name="dran"/>. Она имеет типичную для сегментирующих сетей архитектуру, представленную на рисунке 5, и состоит из нескольких слоев, понижающих степень дискретизации данных, затем нескольких расширяющих слоев с тремя декодерами. | ||
| + | |||
| + | Особенностью данных микроскопии является разный масштаб ядер на изображениях, поэтому в сеть подаются не только исходные изображения, а еще и уменьшенные и увеличенные в 2 раза. Такой подход частично решает проблему влияния существенно различающегося масштаба и получил название многомасштабной сети глубокой остаточной агрегации (англ. Multiscale Deep Residual Aggregation Network, MDRAN)<ref name="dran"/>. | ||
| + | |||
| + | == Детекция клеток == | ||
| + | Во многих биологических экспериментах необходимо уметь детектировать клетки, за которыми ведется наблюдение, понимать сколько их, как они расположены относительно друг друга. Для решения этих задач в компьютерном зрении используется несколько разных подходов<ref>[https://ieeexplore.ieee.org/document/5711528 Takeo Kanade {{---}} Cell image analysis: Algorithms, system and applications, 2011]</ref>. Одни используют сверточные сети, чтобы предсказывать карту плотности, другие основаны на построении деревьев максимально устойчивых экстремальных областей. Вне зависимости от реализации, методы детекции клеток направлены на оценку количества клеток и учитывают перекрывания, неравномерность распредления клеток и другие факторы, специфичные для микроскопических изображений. | ||
| + | |||
| + | === Подсчет клеток на основе сверточных сетей === | ||
| + | [[Файл:FCRN-A_and_FCRN_B.png|425px|thumb|right|Рисунок 6. Архитекутры полносверточных регрессионных сетей для построения карт плотности<ref name="fcrn">[https://www.robots.ox.ac.uk/~vgg/publications/2016/Xie16/xie16.pdf Weidi Xie {{---}} Microscopy cell counting and detection with fully convolutional regression networks, 2016]</ref>.]] | ||
| + | К автоматическому подсчету клеток можно подойти с разных сторон. Первый подход основан на детекции с предварительной сегментацией изображения. Процесс сегментации сам по себе сложен и существует более эффективный способ. В его основе лежит регрессия и оценка плотности без непосредственной детекции и сегментации. По карте плотности можно с хорошей точностью оценить количество клеток. | ||
| + | |||
| + | Особенностью изображений микроскопии является то, что клетки в большинстве случаев имеют размер значительно меньший, чем размер изображения, то есть нет необходимости использовать сложные глубокие сети, которые способны выучить высоко семантическую информацию. Поэтому используются полносверточные регрессионные сети (англ. Fully Convolutional Regression Networks, FCRN)<ref name="fcrn"/>. Различия в архитектурах состоят в размерах ядер и количестве слоев (рисунок 6). Такие сети на выходе дают карту плотности клеток. | ||
| + | |||
| + | Такой подход позволяет проводить непрерывное обучение с изображениями произвольных размеров, что важно в том числе для покадровой съемки и изучения длительных процесоов. Он также обеспечивает интуитивное понимание представлений функций из FCRN, визуализируя, в какой степени информация была закодирована на разных уровнях. | ||
| + | |||
| + | === Распознавание перекрывающихся объектов на основе деревьев экстремальных областей === | ||
| + | [[Файл:Extremal Region Trees.png|left|250px|thumb|Рисунок 7. Получающаяся древовидная структура<ref name="tree">[http://sites.skoltech.ru/app/data/uploads/sites/25/2014/11/MIA15.pdf Carlos Arteta {{---}} Detecting Overlapping Instances in Microscopy Images Using Extremal Region Trees, 2016]</ref>.]] | ||
| + | Клетки на изображениях микроскопии могут быть распределены неравномерно, группироваться, перекрываться, что затрудняет подсчет количества клеток и их дальнейшее изучение, так как стандартные методы применимы либо к изображениям с высокой плотностью объектов и не пытаются их разделить, либо наоборот эффективно работают только с изображениями с низкой плотностью. | ||
| + | |||
| + | Чтобы учесть все особенности изображения используется метод, основанный на древовидной дискретной графической модели, которая позволяет выбрать и промаркировать набор непересекающихся участков изображения с помощью глобальной оптимизации<ref name="tree"/>. Каждый регион маркируется в соответствии с количеством объектов, которые он содержит. В условиях низкой плотности объектов метод, как правило, находит и выделяет отдельные клетки, а в местах, где клетки перекрываются, предложенный метод выделяет группы клеток (рисунок 7). Подобное адаптивное поведение, управляемое оптимизационным процессом, является уникальным для данного метода. | ||
| + | |||
| + | Нужно также отметить, что вывод модели эффективен с точки зрения вычислений и требует всего нескольких сотен оценок классификатора, за которыми следует динамическое программирование на поддеревьях. | ||
| + | |||
| + | == Склеивание изображений == | ||
| + | Не всегда изучаемый объект полностью помещается на один снимок микроскопа. В связи с этим стоит задача склеивания изображений для дальнейшего исследования целых клеточных культур, используя различные методы визуализации. Эту задачу может решить инструмент для сшивания изображений микроскопии (англ. Microscopy Image Stitching Tool, MIST)<ref>[https://ieeexplore.ieee.org/document/7359951 Joe Chalfoun {{---}} MIST: Microscopy Image Stitching Tool, 2015]</ref>. Программа предназначена для быстрого и точного склеивания больших двумерных сеток перекрывающихся изображений покадровых снимков микроскопа. В основе работы MIST лежит оценка параметров модели на основе вычисленных попарных перемещений, а затем минимизация ошибок склеивания путем оптимизации перемещений в пределах квадратной области. | ||
| + | [[Файл:MIST.jpg|left|450px|thumb|Рисунок 8. Примеры изображений, склеенных с помощью MIST<ref name="mist">[https://www.nature.com/articles/s41598-017-04567-y Joe Chalfoun {{---}} MIST: Accurate and Scalable Microscopy Image Stitching Tool with Stage Modeling and Error Minimization, 2017]</ref>.]] | ||
| + | Алгоритм MIST состоит из четырех шагов<ref name="mist"/>. Сначала вычисляются всевозможные перемещения между соседними покадровыми изображениями. После чего, если параметры модели не указаны пользователем, оценивается модель механического предметного столика на основе вычисленных перемещений. Затем оптимизируются перемещения между изображениями для уменьшения ошибок склеивания и составляется полученное изображение (рисунок 8). | ||
| + | |||
| + | Необходимо помнить, что MIST был разработан для склеивания микроскопических изображений, полученных с помощью механического предметного столика, который перемещает образец по повторяющейся сетке, что накладывает ограничение на его использование. | ||
| + | |||
| + | == Улучшение качества изображений == | ||
| + | [[Файл:Autofocus cnn.png|leftright|400px|thumb|Рисунок 9. (a) Архитектура сверточной нейронной сети для предсказания положения фокуса микроскопа. | ||
| + | (b) Примеры изображений с разным фокусным расстоянием<ref name="focus">[https://www.nature.com/articles/s41598-018-25458-w/ Ling Wei {{---}} Reconstructing cell cycle and disease progression using deep learning, 2018]</ref>.]] | ||
| + | Зачастую изображения, полученные с помощью микроскопии, не имеют достаточно хорошее для дальнейшей работы качество. Есть разные способы борьбы с этим. Можно улучшать качество двумерных изображений стандартными методами, не имеющих отличий, связанных со специфичностью данных, или же заранее пытаться получить высококачественное изображение, предсказывая положение фокуса. | ||
| + | |||
| + | === Предсказание положения фокуса === | ||
| + | При покадровой съемке длительного непрерывного процесса необходимо постоянно следить за положением фокуса микроскопа, чтобы не получать размытые изображения. Процесс выставления фокуса можно автоматизировать, построив сеть, которая будет предсказывать нужное положение. Эту задачу можно свести к задаче классификации изображений по фокусному расстоянию во время съемки. Для ее решения используется сверточная сеть (рисунок 9), которая состоит из двух блоков свертки и двух полносвязных блоков для классификации<ref name="focus"/>. | ||
| + | |||
| + | Такая сверточная сеть показывает большую точность, чем группа людей-экспертов. По сравнению с другими подходами к автофокусировке, сеть не требует физической калибровки и устойчива к шуму, оптическим артефактам и другим особенностям. | ||
| + | |||
| + | === Восстановление рассеянных трехмерных изображений === | ||
| + | [[Файл:3d restoration net.jpeg|right|400px|thumb|Рисунок 10. Схема работы архитектуры ScatNet<ref name="scatnet">[https://www.osapublishing.org/oe/fulltext.cfm?uri=oe-28-20-30234&id=439993 Le Xiao {{---}} Deep learning-enabled efficient image restoration for 3D microscopy of turbid biological specimens, 2020]</ref>.]] | ||
| + | Трехмерная флуоресцентная микроскопия является важным инструментом для современных исследований, но ее более широкому применению препятствует рассеяние света биологическими образцами. В основе подхода, который способен восстановить размытое и рассеянное светом трехмерное изображение глубоких тканей, лежит сеть ScatNet<ref>[https://www.di.ens.fr/data/software/scatnet/ ScatNet: homepage of the project]</ref>. | ||
| + | |||
| + | В течение каждой эпохи обучения ScatNet учится, как лучше восстанавливать высококачественные изображения из размытых входных данных. Сгенерированный промежуточный результат для каждой эпохи сравнивается с данными фиксированной метки для оптимизации функции потерь сети<ref name="scatnet"/>. Более детальная схема работы представлена на рисунке 10. ScatNet способствует простому и быстрому восстановлению изображений и не требует трудоемких ручных операций. | ||
| + | |||
| + | Такой подход позволяет с помощью вычислений увеличить глубину визуализации изображений трехмерной флуоресцентной микроскопии без добавления сложной оптики или моделей оптического рассеяния, так как основан только на мощных возможностях прогнозирования сверточной нейронной сети. | ||
| − | == | + | = См. также = |
| + | * [[ Компьютерное зрение ]] | ||
| + | * [[ Глубокое обучение ]] | ||
| + | * [[ Сверточные нейронные сети ]] | ||
| + | * [[ Batch-normalization ]] | ||
| + | * [[ Сегментация изображений ]] | ||
| − | == | + | = Примечания = |
| − | + | <references/> | |
| − | |||
| − | |||
| − | + | = Источники информации = | |
| + | * [https://ru.wikipedia.org/wiki/%D0%A4%D0%BB%D1%83%D0%BE%D1%80%D0%B5%D1%81%D1%86%D0%B5%D0%BD%D1%82%D0%BD%D0%B0%D1%8F_%D0%BC%D0%B8%D0%BA%D1%80%D0%BE%D1%81%D0%BA%D0%BE%D0%BF%D0%B8%D1%8F Википедия: Флуоресцентная микроскопия] | ||
| + | * [https://www.nature.com/articles/d41586-020-01128-8 Deep learning takes on tumours] | ||
| + | * [https://towardsdatascience.com/deep-learning-on-microscopy-imaging-865b521ec47c Deep Learning on Microscopy Imaging] | ||
| + | * [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7075610/ Leonhard Möckl {{---}} Deep learning in single-molecule microscopy: fundamentals, caveats, and recent developments, 2020] | ||
| + | * [https://habr.com/ru/company/skillfactory/blog/528242/ Реконструкция нейронных карт по данным электронной микроскопии с помощью глубокого обучения] | ||
| + | * [https://nplus1.ru/news/2017/09/08/jurkat-cells Глубокое обучение помогло визуализировать биологические процессы] | ||
| + | * [https://nplus1.ru/news/2018/04/17/arm Микроскоп с нейросетью и дополненной реальностью упростит обнаружение рака] | ||
| + | * Mei Chen {{---}} Computer Vision for Microscopy Image Analysis, 1st Edition, 2019 | ||
| + | * Бхарат Рамсундар {{---}} Глубокое обучение в биологии и медицине, 2020, C. 99-122 | ||
[[Категория: Машинное обучение]] | [[Категория: Машинное обучение]] | ||
[[Категория: Компьютерное зрение]] | [[Категория: Компьютерное зрение]] | ||
| + | [[Категория: Практическое применение машинного обучения ]] | ||
Текущая версия на 19:27, 4 сентября 2022
Компьютерное зрение помогает автоматизировать обработку изображений, полученных с помощью микроскопии. С его появлением стало возможным эффективно и с хорошей точностью классифицировать клетки, сегментировать полученные изображения, улучшать их качество и решать другие задачи без участия человека[1].
Задачи компьютерного зрения в микроскопии
На данный момент компьютерное зрение нашло применение в большинстве направлений, где есть необходимость обрабатывать и анализировать изображения. Микроскопия не стала исключением. Теперь задачи, направленные непосредственно на работу с изображениями, можно решить используя методы глубокого обучения, например, построить подходящую сверточную нейронную сеть[2].
Классификация клеток
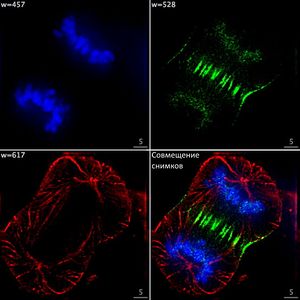
Классификация клеток является базовой задачей микроскопии. Обычно для этого используются изображения, полученные на флуоресцентных микроскопах (рисунок 1), так как классификаторы для изображений с обычных оптических микроскопов не способны отразить биологическое разнообразие различных типов клеток. Клетки можно делить по фазе в клеточном цикле, типу (повержденные или нет, раковые или нормальные), физиологическому состоянию, виду и другим признакам. Для большинства задач классификации уже существуют готовые архитектуры сверточных сетей[4][5][6][7][8][9][10].
Определение фазы клеточного цикла
Одним из признаков, по которым можно разделить клетки, является определение фазы клеточного цикла, в которой находится клетка. Эта задача имеет практическое применение для обнаружения поврежденных клеток, которые при визуализации будут кластеризоваться отдельно от остальных. Сверточная сеть для решения задачи изображена на рисунке 2. Она обучается на изображениях с флуоресцентными метками, о которых было сказано ранее, и дает на выходе не только классификацию каждой клетки, а также визуализирует процесс клеточного цикла, используя нелинейное уменьшение размерности[5]. Классификация и визуализация являются всего лишь различными способами интерпретации результатов, поэтому строятся на основе одних и тех же выведенных закономерностей.
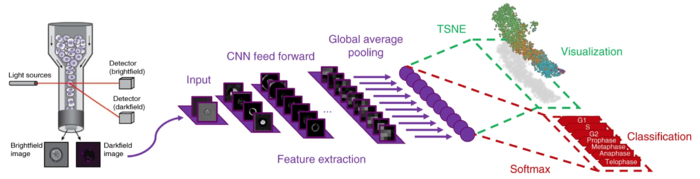
Особенностью работы описанной сверточной сети является необходимость разметить только небольшую часть данных, на основании чего она далее учится размечать самостоятельно. Интересно, что при визуализации фазы становятся упорядочены в хронологически правильном порядке, несмотря на то, что информация о порядке фаз не передавалась в сеть напрямую. Это говорит об эффективности использования флуоресцентных меток в качестве категориальных.
Идентификация раковых клеток
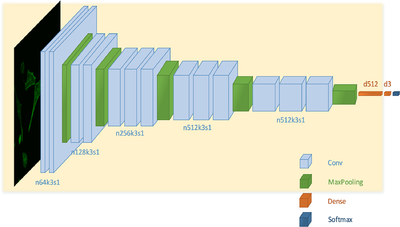
Другой задачей классификации является обнаружение раковых клеток. Для ее решения используется сверточная нейронная сеть, изображенная на рисунке 3. Она имеет архитектуру VGG-16, в которой дополнительно после каждой функции активации добавлена пакетная нормализация для регуляризации[6]. Сеть, как и остальные классификаторы изображений микроскопии, принимает на вход изображения с флуоресцентными метками.
Особенностью при обучении представленной сети является использование из-за недостаточного объема данных трансферного обучения, то есть модель предварительно обучается на другом огромном объеме данных. В данном случае первые 14 слоев сначала обучаются на наборе данных классификации ImageNet, а потом уже происходит обучение для классификации раковых и нормальных клеток. Такая сверточная сеть лучше справляется с задачей классификации клеток по сравнению с экспертом-человеком, особенно на изображениях с недостаточно хорошим качеством.
Классификация лейкоцитов
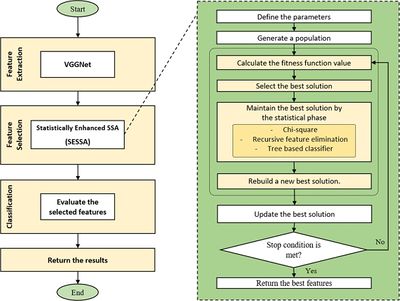
Эта задача отличается от предыдущих тем, что не требует предварительной обработки материалов и использования флуоресцентного микроскопа, для ее решения достаточно изображений с оптического микроскопа. Лейкоциты важно уметь классифицировать, потому что они играют значительную роль в организме человека (иногда в крови могут образоваться злокачественные лейкоциты, которые способны вызывать лейкемию). Автоматическая классификация лейкоцитов может помочь специалистам в лабораториях, где нет достаточного количества квалифицированных сотрудников для проведения анализа крови.
Для решения задачи используется сеть VGGNet, похожая на ту, что идентифицирует раковые клетки, и так же трансферное обучение. Отличительной деталью является использования для оптимизации статистически усиленного алгоритма сальпового роя (англ. Statistically Enhanced Salp Swarm Algorithm, SESSA)[7] для выбора наиболее важных функций, извлеченных с помощью сети VGGNet, и удаления сильно коррелированных и шумных функций. Общая структура алгоритма представлена на рисунке 4. Такая оптимизация позволяет сократить время обучения, повысить точность и избавиться от переобучения. Результаты этого гибридного подхода к классификации являются самыми эффективными среди всех известных опубликованных работ по тому же набору данных.
Сегментация изображений
Задача сегментации изображений, полученных с микроскопа, состоит в том, чтобы аннотировать их, то есть отмечать границы объектов (клеток, ядер). Имея сегментированное изображение, легче проводить дальнейший анализ и изучать конкретные части клетки[11].
Сеть U-Net
Для решения задачи сегментации обычно используется модифицированная полносвязная сверточная сеть U-Net[13]. Сеть U-Net получила широкое распространение благодаря способности последовательно распознавать как большие, так и мелкие частицы, а также устойчивости к различным условиям визуализации и наборам данных. Также она показывает хорошие результаты, даже если размер набора даных для обучения небольшой, что является частой проблемой анализа изображений, полученных с микроскопа. Однако минусом сети U-Net является ограничение на размер входного изображения, в то время как разрешение микроскопических изображений только растет с течением времени.
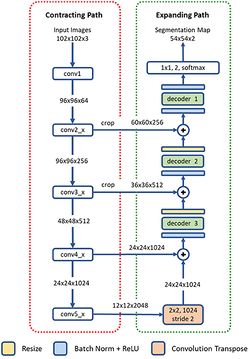
Сегментация ядер клеток
Сеть U-Net хоть и является универсальной, но для решения некоторых задач удобнее использовать более специализированные сети. Для задачи точной сегментации ядер клеток строится сеть глубокой остаточной агрегации (англ. Deep Residual Aggregation Network, DRAN)[12]. Она имеет типичную для сегментирующих сетей архитектуру, представленную на рисунке 5, и состоит из нескольких слоев, понижающих степень дискретизации данных, затем нескольких расширяющих слоев с тремя декодерами.
Особенностью данных микроскопии является разный масштаб ядер на изображениях, поэтому в сеть подаются не только исходные изображения, а еще и уменьшенные и увеличенные в 2 раза. Такой подход частично решает проблему влияния существенно различающегося масштаба и получил название многомасштабной сети глубокой остаточной агрегации (англ. Multiscale Deep Residual Aggregation Network, MDRAN)[12].
Детекция клеток
Во многих биологических экспериментах необходимо уметь детектировать клетки, за которыми ведется наблюдение, понимать сколько их, как они расположены относительно друг друга. Для решения этих задач в компьютерном зрении используется несколько разных подходов[14]. Одни используют сверточные сети, чтобы предсказывать карту плотности, другие основаны на построении деревьев максимально устойчивых экстремальных областей. Вне зависимости от реализации, методы детекции клеток направлены на оценку количества клеток и учитывают перекрывания, неравномерность распредления клеток и другие факторы, специфичные для микроскопических изображений.
Подсчет клеток на основе сверточных сетей
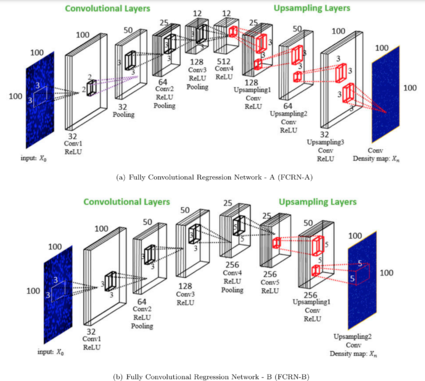
К автоматическому подсчету клеток можно подойти с разных сторон. Первый подход основан на детекции с предварительной сегментацией изображения. Процесс сегментации сам по себе сложен и существует более эффективный способ. В его основе лежит регрессия и оценка плотности без непосредственной детекции и сегментации. По карте плотности можно с хорошей точностью оценить количество клеток.
Особенностью изображений микроскопии является то, что клетки в большинстве случаев имеют размер значительно меньший, чем размер изображения, то есть нет необходимости использовать сложные глубокие сети, которые способны выучить высоко семантическую информацию. Поэтому используются полносверточные регрессионные сети (англ. Fully Convolutional Regression Networks, FCRN)[15]. Различия в архитектурах состоят в размерах ядер и количестве слоев (рисунок 6). Такие сети на выходе дают карту плотности клеток.
Такой подход позволяет проводить непрерывное обучение с изображениями произвольных размеров, что важно в том числе для покадровой съемки и изучения длительных процесоов. Он также обеспечивает интуитивное понимание представлений функций из FCRN, визуализируя, в какой степени информация была закодирована на разных уровнях.
Распознавание перекрывающихся объектов на основе деревьев экстремальных областей
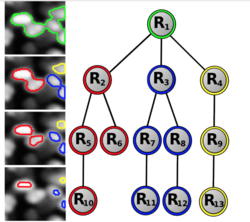
Клетки на изображениях микроскопии могут быть распределены неравномерно, группироваться, перекрываться, что затрудняет подсчет количества клеток и их дальнейшее изучение, так как стандартные методы применимы либо к изображениям с высокой плотностью объектов и не пытаются их разделить, либо наоборот эффективно работают только с изображениями с низкой плотностью.
Чтобы учесть все особенности изображения используется метод, основанный на древовидной дискретной графической модели, которая позволяет выбрать и промаркировать набор непересекающихся участков изображения с помощью глобальной оптимизации[16]. Каждый регион маркируется в соответствии с количеством объектов, которые он содержит. В условиях низкой плотности объектов метод, как правило, находит и выделяет отдельные клетки, а в местах, где клетки перекрываются, предложенный метод выделяет группы клеток (рисунок 7). Подобное адаптивное поведение, управляемое оптимизационным процессом, является уникальным для данного метода.
Нужно также отметить, что вывод модели эффективен с точки зрения вычислений и требует всего нескольких сотен оценок классификатора, за которыми следует динамическое программирование на поддеревьях.
Склеивание изображений
Не всегда изучаемый объект полностью помещается на один снимок микроскопа. В связи с этим стоит задача склеивания изображений для дальнейшего исследования целых клеточных культур, используя различные методы визуализации. Эту задачу может решить инструмент для сшивания изображений микроскопии (англ. Microscopy Image Stitching Tool, MIST)[17]. Программа предназначена для быстрого и точного склеивания больших двумерных сеток перекрывающихся изображений покадровых снимков микроскопа. В основе работы MIST лежит оценка параметров модели на основе вычисленных попарных перемещений, а затем минимизация ошибок склеивания путем оптимизации перемещений в пределах квадратной области.

Алгоритм MIST состоит из четырех шагов[18]. Сначала вычисляются всевозможные перемещения между соседними покадровыми изображениями. После чего, если параметры модели не указаны пользователем, оценивается модель механического предметного столика на основе вычисленных перемещений. Затем оптимизируются перемещения между изображениями для уменьшения ошибок склеивания и составляется полученное изображение (рисунок 8).
Необходимо помнить, что MIST был разработан для склеивания микроскопических изображений, полученных с помощью механического предметного столика, который перемещает образец по повторяющейся сетке, что накладывает ограничение на его использование.
Улучшение качества изображений
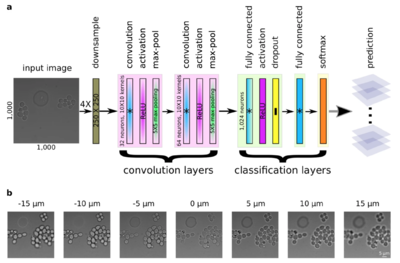
Зачастую изображения, полученные с помощью микроскопии, не имеют достаточно хорошее для дальнейшей работы качество. Есть разные способы борьбы с этим. Можно улучшать качество двумерных изображений стандартными методами, не имеющих отличий, связанных со специфичностью данных, или же заранее пытаться получить высококачественное изображение, предсказывая положение фокуса.
Предсказание положения фокуса
При покадровой съемке длительного непрерывного процесса необходимо постоянно следить за положением фокуса микроскопа, чтобы не получать размытые изображения. Процесс выставления фокуса можно автоматизировать, построив сеть, которая будет предсказывать нужное положение. Эту задачу можно свести к задаче классификации изображений по фокусному расстоянию во время съемки. Для ее решения используется сверточная сеть (рисунок 9), которая состоит из двух блоков свертки и двух полносвязных блоков для классификации[19].
Такая сверточная сеть показывает большую точность, чем группа людей-экспертов. По сравнению с другими подходами к автофокусировке, сеть не требует физической калибровки и устойчива к шуму, оптическим артефактам и другим особенностям.
Восстановление рассеянных трехмерных изображений
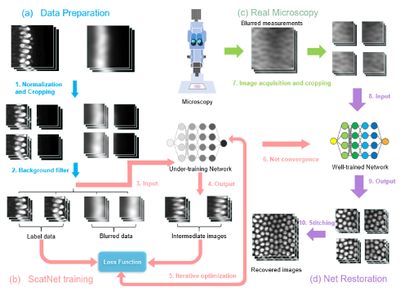
Трехмерная флуоресцентная микроскопия является важным инструментом для современных исследований, но ее более широкому применению препятствует рассеяние света биологическими образцами. В основе подхода, который способен восстановить размытое и рассеянное светом трехмерное изображение глубоких тканей, лежит сеть ScatNet[21].
В течение каждой эпохи обучения ScatNet учится, как лучше восстанавливать высококачественные изображения из размытых входных данных. Сгенерированный промежуточный результат для каждой эпохи сравнивается с данными фиксированной метки для оптимизации функции потерь сети[20]. Более детальная схема работы представлена на рисунке 10. ScatNet способствует простому и быстрому восстановлению изображений и не требует трудоемких ручных операций.
Такой подход позволяет с помощью вычислений увеличить глубину визуализации изображений трехмерной флуоресцентной микроскопии без добавления сложной оптики или моделей оптического рассеяния, так как основан только на мощных возможностях прогнозирования сверточной нейронной сети.
См. также
Примечания
- ↑ Gaudenz Danuser — Computer Vision in Cell Biology, 2011
- ↑ Nikita V Orlov — Computer Vision for Microscopy Applications, 2007
- ↑ Википедия: Флуоресценция в биологических исследованиях
- ↑ Håkan Öhrn — General image classifier for fluorescence microscopy using transfer learning , 2019
- ↑ 5,0 5,1 5,2 Philipp Eulenberg — Reconstructing cell cycle and disease progression using deep learning, 2017
- ↑ 6,0 6,1 6,2 Ronald Wihal Oei — Convolutional neural network for cell classification using microscope images of intracellular actin networks, 2019
- ↑ 7,0 7,1 7,2 Ahmed T. Sahlol — Efficient Classification of White Blood Cell Leukemia with Improved Swarm Optimization of Deep Features, 2020
- ↑ Samuel Berryman — Image-based Cell Phenotyping Using Deep Learning, 2019
- ↑ Nan Meng — Computational single-cell classification using deep learning on bright-field and phase images, 2017
- ↑ Sachin Mehta — Y-Net: Joint Segmentation and Classification for Diagnosis of Breast Biopsy Images, 2018
- ↑ James P. Horwath — Understanding important features of deep learning models for segmentation of high-resolution transmission electron microscopy images, 2020
- ↑ 12,0 12,1 12,2 Quoc Dang Vu — Methods for Segmentation and Classification of Digital Microscopy Tissue Images, 2019
- ↑ Olaf Ronneberger— Reconstructing cell cycle and disease progression using deep learning, 2015
- ↑ Takeo Kanade — Cell image analysis: Algorithms, system and applications, 2011
- ↑ 15,0 15,1 Weidi Xie — Microscopy cell counting and detection with fully convolutional regression networks, 2016
- ↑ 16,0 16,1 Carlos Arteta — Detecting Overlapping Instances in Microscopy Images Using Extremal Region Trees, 2016
- ↑ Joe Chalfoun — MIST: Microscopy Image Stitching Tool, 2015
- ↑ 18,0 18,1 Joe Chalfoun — MIST: Accurate and Scalable Microscopy Image Stitching Tool with Stage Modeling and Error Minimization, 2017
- ↑ 19,0 19,1 Ling Wei — Reconstructing cell cycle and disease progression using deep learning, 2018
- ↑ 20,0 20,1 Le Xiao — Deep learning-enabled efficient image restoration for 3D microscopy of turbid biological specimens, 2020
- ↑ ScatNet: homepage of the project
Источники информации
- Википедия: Флуоресцентная микроскопия
- Deep learning takes on tumours
- Deep Learning on Microscopy Imaging
- Leonhard Möckl — Deep learning in single-molecule microscopy: fundamentals, caveats, and recent developments, 2020
- Реконструкция нейронных карт по данным электронной микроскопии с помощью глубокого обучения
- Глубокое обучение помогло визуализировать биологические процессы
- Микроскоп с нейросетью и дополненной реальностью упростит обнаружение рака
- Mei Chen — Computer Vision for Microscopy Image Analysis, 1st Edition, 2019
- Бхарат Рамсундар — Глубокое обучение в биологии и медицине, 2020, C. 99-122