Машинное обучение в медицине
Машинное обучение в медицине используют очень активно, находя все больше областей для его применения. Сейчас в медицине используют почти все виды машинного обучения: обучение с учителем, без учителя, с частичным привлечением учителя, с подкреплением. Наиболее активно машинное обучение в медицине используют для решения проблем по диагностике заболеваний и дизайну лекарств. Эти задачи относят к обучению с учителем или с частичным привлечением учителя. Кроме того, машинное обучение стали применять в персонализированной медицине и генерации данных различных исследований для анонимизации данных пациентов. В этих задачах сейчас все больше применяют обучение с подкреплением и обучение без учителя, в частности, генеративные состязательные сети. Машинное обучение в медицине начали изучать еще в 2000-ых и оно продолжает активно развиваться и в наши дни.
Диагностика заболеваний по результатам рентгенологических и УЗИ исследований
Диагностика по изображению
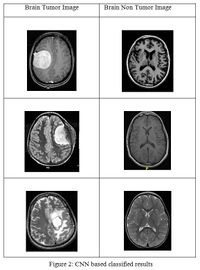

В диагностике заболеваний есть большое количество задач, которые можно решить при помощи машинного обучения, а в частности, при помощи анализа результатов различных исследований, таких как рентген, УЗИ или МРТ. В основном задача любой модели сводится к предсказанию, болен ли человек сейчас. В зависимости от целей исследования используют разные типы классификации: обычную (рис. 1), мультиклассовую или вероятностную (рис. 2).
Для решения таких задач чаще всего используют глубокое обучение. Такие модели на вход получают картинку с рентгенологическим или ультразвуковым исследованием пациента и по ним предсказывают наличие болезни. Обычно внутри таких моделей-классификаторов лежат сверточные нейронные сети, а иногда к ним добавляется механизм внимания. За основу берутся state-of-the-art модели в области сверточных нейронных сетей, такие как GoogLeNet[3], при этом точность предсказаний превышает 90%. Такие модели учатся на размеченных тренировочных наборах данных, поэтому их можно отнести к обучению с учителем. Большое распространение такие классификаторы получили в предсказании злокачественности новообразований, классификации заболеваний легких, подборе дозы контраста при проведении МРТ.
Применения

Многие модели учатся не только определять, есть ли опухоль на данном образце, но и локализировать ее (рис. 3). Таким образом, необходимо решить задачу сегментации изображения, то есть выделения каких-то ее наиболее важных частей. Технически задача не сильно отличается от предыдущей и решается все теми же сверточными нейронными сетями.
Поскольку точность предсказаний у описанных выше моделей достаточно высока, их стали применять на практике. Сейчас ведутся исследования по внедрению таких моделей в УЗИ-аппараты для того, чтобы быстрее и точнее определять местоположение и злокачественность опухоли. Кроме того, такие модели стали применять в направленной лучевой терапии, когда злокачественная опухоль облучается различными видами частиц. Известно, что эти частицы уничтожают не только раковые, но и здоровые клетки. Именно поэтому активно внедряются модели, которые могут подсказать аппарату точное направление облучения. Также сверточные сети стали использовать для определения дозы контрастного вещества при МРТ[5].
Генерация результатов исследований
Для обучения сверточных нейронных сетей необходимо большое количество данных, которые очень часто достаточно тяжело или даже невозможно получить из-за запрета на использование данных исследований без согласия пациента. Поэтому сейчас для получения достаточно больших датасетов стали применять генеративные состязательные сети.
Задачу таких GAN можно разделить на две: генерация результатов исследований здоровых пациентов и пациентов с патологиями. В случае второй задачи важно, чтобы сгенерированные изображения правильно определялись именно по типу патологии.
Основная проблема, с которой сталкиваются такие модели — необходимость очень точно определять границы объекта на сгенерированном изображении, а также не допускать размытости. Эти две проблемы долгое время не получалось решить без большого количества реальных данных, вследствие чего не было возможным применение сгенерированных изображений на практике.
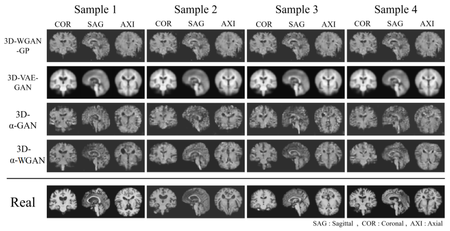
Сильно улучшить поведение моделей удалось путем использования метрики Васерштейна[на 09.01.21 не создан]. Интуитивно, если каждая мера измеряет распределение «грунта» по метрическому пространству, то расстояние Васерштейна измеряет минимальную стоимость преобразования одного распределения грунта в другое, при этом предполагается, что стоимость прямо пропорциональна количеству грунта и расстоянию, на которое его надо перетащить. Использование такой метрики в GAN помогло сильно улучшить поведение моделей при генерации данных МРТ исследований. На рисунке 4 в четвертой строке показаны изображения, сгенерированные при помощи сети, использующей метрику Васерштейна.
Персонализированная медицина
Персонализированная медицина (англ. personalized medicine) — новая организационная модель построения медицинской помощи пациентам, которая основывается на подборе индивидуальных лечебных, диагностических и превентивных средств, оптимально подходящих по биохимическим, физиологическим и генетическим особенностям организма.
Основная цель нового направления в медицине заключается в персонализации и оптимизации профилактических мероприятий и лечения пациентов для исключения негативных последствий и осложнений, проявляющихся из-за индивидуальных особенностей. Основные отрасли медицины, где применяются новые принципы — онкология, фармация и фармакогеномика. Последняя занимается изучением реакций организма на медицинские препараты в зависимости от индивидуальных наследственных факторов.
Экспрессия генов и анализ транскриптомных данных

Многие модели ориентируются на данные экспрессии генов[8] (в широком смысле — процесс получения белка из последовательности ДНК). Известно, что от количества некоторых белков напрямую зависит возможность клеток становиться раковыми, а также порождать другие заболевания. Совокупность изменений в большом количестве различных белков может приводить к заболеванию. Именно поэтому модели персонализированной медицины основываются на данных экспрессии. Часто в качестве основы используют сверточные нейронные сети (рис. 5), располагая гены, отвечающие за похожие по своей функции белки, рядом друг с другом.
Прямой анализ экспрессии генов — трудная и дорогостоящая задача, поэтому часто обращаются к транскриптомным данным. Транскриптом — совокупность всех молекул РНК[9], которые присутствовали в клетке после завершения процесса трансрипции (получение РНК с матрицы ДНК). Транскриптом включает в себя матричную РНК (мРНК) — РНК, с которой впоследствии транслируются белки, и некодирующую РНК, которая, в свою очередь, не используется для получения белков. Транскриптомные данные чаще всего получают с помощью РНК-секвенирования (англ. RNA-seq) или ДНК-микрочипов (англ. DNA-microarray). С помощью специальных процедур из взятой у пациента пробы выделяют мРНК, которая затем наносится на ДНК-микрочип, где цепочки мРНК зацепляются, образуя двухцепочечные молекулы. Число цепочек, зацепившихся за определенный участок ДНК-микрочипа, определяет интенсивность свечения этого участка при сканировании. Так косвенно определяют экспрессию каждого из генов. Следующая проблема, с которой сталкиваются исследователи — слишком большое количество генов (например, у человека их около 28000). Вследствие этого очень тяжело обучать модели, поэтому приходится прибегать к уменьшению размерности.
Уменьшение размерности при работе с данными экспрессии генов

Для уменьшения размерности в случае биологических данных применяются методы, основным критерием которых становится их скорость. Поскольку количество генов очень велико, чаще всего нет возможности опробовать встроенные и оберточные методы. Чаще всего, используют фильтры.
Одним из примеров моделей, основанных на фильтрах является модель MeLiF[11] (рис. 6), которая берет несколько фильтров с соответствующими функциями измерения релевантности признака , после чего строит новую меру значимости как , где — набор вещественных коэффициентов. После получения новой меры значимости происходит оценка модели на лучших выбранных признаках. В случае удовлетворительного результата алгоритм завершается, возвращая текущий набор признаков, иначе — возвращается снова к получению новой меры на признаках.
Медицинская карта
Другая задача — предсказать, заболеет ли человек в будущем. Для этого часто используется электронная медицинская карта пациента, и на вход сети подаются данные о пациенте, результаты исследований, его генеалогическое древо и другая медицинская информация. Основываясь на полученных данных, модель должна предсказать вероятность того, что человек может заболеть каждой болезнью из приведенного набора. Если вероятность хотя бы по одному из маркеров превышает некоторое значение — такая модель рекомендует пациенту обратиться к специалисту и относит его в группу высокого риска по этому параметру. В свою очередь специалист может подтвердить или же опровергнуть опасения модели, отдавая ей результат — права модель или нет. Таким образом, эта проблема относится к классу задач обучения с подкреплением. С точки зрения архитектуры моделей — в таких задачах часто используют метод опорных векторов (SVM) или глубокое обучение.
Поиск лекарств
Поиск лекарств (англ. drug discovery, drug design) — процесс поиска новых лекарственных средств, часто основанный на знаниях о биологической мишени (целевом белке, с которым должно взаимодействовать лекарственное вещество). При поиске новых лекарственных средств часто прибегают к помощи машинного обучения в таких задачах, как предсказание молекулярных свойств потенциальной молекулы лекарства, формы какого-либо белка, активности взаимодействия между веществами.
Предсказание молекулярных свойств
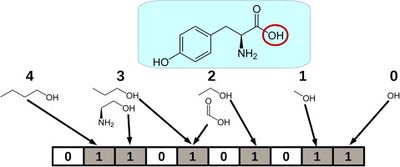
Одна из главных задач машинного обучения при поиске новых лекарств — сужение круга их поиска. Чаще всего фармацевтические компании имеют на руках библиотеки с огромным количеством веществ, которые они потенциально могут синтезировать и опробовать в качестве лекарства. Но обычно размеры этих библиотек составляют тысячи молекул, поэтому синтезировать и проверить каждую из них не представляется возможным. В этом случае прибегают к предсказанию некоторых свойств этих молекул, которые точно определяют, может ли молекула быть использована как лекарство. Для предсказания свойств молекул обычно используют молекулярный граф — графическое представление молекулы (ее атомов и связей). Пример молекулярного графа можно увидеть на рисунке 7.
Первые нейронные сети для предсказания молекулярных свойств использовали Моргановские фингерпринты (англ. Morgan fingerpints), которые для каждого атома в молекуле выделяли всех его соседей на каком-то определенном расстоянии (которое является гиперпараметром) и смотрели на наличие такой подструктуры в молекуле (рис. 8). Получался аналог некоторого оne-hot кодирования. Впоследствии этот метод был несколько усовершенствован, и стали смотреть не на наличие подструктуры, а на то, сколько раз она встречается в молекуле. Прорыв в этой области случился с появлением сети NeuralFingerprints [13], которая является примером одной из первых попыток применения графовых нейронных сетей в этой области.
NeuralFingerprints принимает один гиперпараметр — максимальное расстояние, которое нужно учитывать при просмотре соседей каждого атома. После этого для каждого расстояния для каждого атома суммируются атомные представления его соседей на текущем расстоянии. Таким образом, получается векторное представление текущего атома фиксированной длины, которая равна количеству свойств у одного атома. Каждый элемент такого представления умножается на обучаемый параметр, уникальный для номера свойства и текущего рассматриваемого расстояния. После этого применяется функция активации к полученному ранее вектору, умноженному на вес, отвечающий за текущее расстояние. Полученные результаты для каждого из атомов на каждом из расстояний суммируются и получается результирующий вектор свойств для молекулы. Таким образом, в этой сети обучаемые параметры — веса для каждого из свойств атомов на каждом расстоянии (, где — индекс номера свойства (в модели их всего 5), — индекс для текущего расстояния) и веса для каждого из расстояний (, где — индекс для текущего расстояния). Псевдокод представлен ниже.
function neuralFingerptint(molecule, R, H, W): # R - максимальное расстояние, H - матрица весов размера len(molecule)R, W - вектор весов размера R f = array[len(molecule), 0] for a in molecule: r[a] = g(a) # записываем свойства для каждого атома for L = 1 to R: for a in molecule: neighbors = neighbors(a) # смотрим соседей на расстоянии L от текущего атома a v = r[a] + sum(r[i] for i in neighbors) # суммируем вектора соседей и вектор текущего атома r[a] = σ(v H[a][L]) # изменяем представление текущего атома i = softmax(r[a] W[L]) # получаем вектор для текущего атома и расстояния f = f + i # добавляем его к ответу return f
Эта сеть была одной из первых в этой области, и сейчас используется как базовый метод в множестве статей. В основу новых методов и сейчас чаще всего ложатся графовые нейронные сети. Подходы графовых нейронных сетей адаптируют под молекулярный граф путем поиска элементов на расстоянии не более, чем N (где N является гиперпараметром), или последовательным рассмотрением каждой вершины и усреднением полученных значений.
Предсказание формы белка

Секвенирование — процесс получения нуклеотидной последовательности из молекулы ДНК. [15] Из нуклеотидной последовательности можно получить аминокислотную последовательность, которая, в свою очередь, кодирует любой белок в организме.
Предсказание формы белка — другая очень важная задача машинного обучения в фармацевтике. С возникновением технологий секвенирования ДНК у сообщества появилась возможность быстро и достаточно качественно прочитывать белковые последовательности, но получать пространственную структуру полученных белков экспериментально все еще очень трудоемко и дорого. Тем не менее, знать пространственную структура белка очень важно, поскольку от нее сильно зависят типы соединений, которые могут связаться (прореагировать) с этим белком. Например, при подборе потенциальной вакцины, необходимо точно знать форму антитела, чтобы понимать, сможет ли это соединение захватить мишень (вирус, бактерию, белок).
Предсказание формы белков основано на нейронных сетях, которые как вход используют очень длинную аминокислотную последовательность (размер алфавита ограничен — всего различных аминокислот 20), а на выходе должны предсказать значения торсионных углов[16] между аминокислотами. В декабре 2020 года DeepMind (исследовательское подразделение Google) заявили[17], что решили проблему пространственной структуры белка. Краткое графическое представление архитектуры AlphaFold2 можно увидеть на рисунке 9. Авторы модели утверждают, что значение метрики глобального расстояния (англ. global distance test) превысило 90%. Метрика глобального расстояния — это метрика, которая вычисляется для каждой аминокислоты как процент атомов углерода из главной цепи белка, которые в сгенерированном белке расположены не более чем на расстоянии какого-то определенного количества ангстрем от соответствующего атома углерода в исходном белке. Обычно это значение берется равным 1, 2, 4 или 8 ангстрем (10-10м).
Генерация молекулярных структур

Еще одна задача, которая есть сейчас в мире машинного обучения — генерировать новые молекулы, которые могут потенциально быть лекарствами. Для этого используют генеративные состязательные сети. Пример генеративной состязательной сети, решающей задачу генерации молекул можно увидеть на рисунке 10. Основное преимущество такого подхода заключается в том, что при работе с библиотеками уже синтезированных лекарств есть вероятность "пропустить" важное соединение просто потому, что в этой библиотеке его не было. Поэтому используется абсолютно другой подход — предлагается наоборот генерировать различные молекулы, а уже потом проверять, действительно ли их можно использовать как лекарство и насколько сложно их синтезировать. Часто эти свойства вносят в метрику качества генератора.
SMILES — способ однозначного задания молекулы при помощи строки. Таким образом, задача генератора — сгенерировать такую строку SMILES для молекулы, чтобы дискриминатор не отличил ее от настоящей. Архитектура дискриминатора чаще всего остается очень похожей на архитектуру обычной сети, предсказывающей молекулярные свойства. Таким образом, в качестве дискриминатора часто используются графовые или сверточные нейронные сети. Для генератора же часто используют механизм памяти, чтобы сеть помнила, какие части уже были сгенерированы и отталкивалась от них.
См. также
Примечания
Источники информации
- ↑ Seetha J, Raja S. S. Brain Tumor Classification Using Convolutional Neural Networks. Biomed Pharmacol J 2018;11(3).
- ↑ CoroNet: A deep neural network for detection and diagnosis of COVID-19 from chest x-ray images, 2020
- ↑ Szegedy et al., Going Deeper with Convolutions, 2015. GoogLeNet.
- ↑ Zhiyun Xue et al., Gender Detection from Spine X-ray Images Using Deep Learning, 2018
- ↑ Lundervold et al., An overview of deep learning in medical imaging focusing on MRI, 2019
- ↑ Generation of 3D Brain MRI Using Auto-Encoding Generative Adversarial Networks
- ↑ Convolutional neural network models for cancer type prediction based on gene expression
- ↑ Википедия: Экспрессия генов
- ↑ Википедия: РНК
- ↑ Сметанников Иван Борисович, Метод и алгоритмы выбора признаков в предсказательном моделировании фенотипических характеристик на основе транскриптомных данных
- ↑ Smetannikov et al., MeLiF: Filter Ensemble Learning Algorithm for Gene Selection
- ↑ Adrià Cereto-Massagué et al., Molecular fingerprint similarity search in virtual screening, 2015
- ↑ Duvenaud et al., Convolutional Networks on Graphs for Learning Molecular Fingerprints, 2015
- ↑ Wikipedia: AlphaFold
- ↑ Википедия: Секвенирование
- ↑ Торсионные углы
- ↑ Фундаментальная «проблема белка» решена
- ↑ Evgeny Putin et al., Reinforced Adversarial Neural Computer for de Novo Molecular Design, 2018