Компьютерное зрение в микроскопии
Компьютерное зрение помогает автоматизировать обработку изображений, полученных с помощью микроскопии. С его появлением стало возможным эффективно и с хорошей точностью классифицировать клетки, сегментировать полученные изображения, улучшать их качество и решать другие задачи без участия человека.
Задачи компьютерного зрения в микроскопии
На данный момент компьютерное зрение нашло применение в большинстве направлений, где есть необходимость обрабатывать и анализировать изображения. Микроскопия не стала исключением. Теперь задачи, направленные непосредственно на работу с изображениями, можно решить, например, построив соответствующую сверточную нейронную сеть.
Классификация клеток
Классификация клеток является базовой задачей микроскопии. Обычно для этого используются изображения, полученные на флуоресцентных микроскопах, так как классификаторы для изображений с обычных оптических микроскопов не способны отразить биологическое разнообразие различных типов клеток. Клетки можно делить по фазе в клеточном цикле, типу (повержденные или нет, раковые или номральные), физиологическому состоянию, виду и другим признакам. Для большинства уже существуют готовые архитектуры сверточных сетей, использующие флуоресцентные метки в качестве категориальных.
Определение фазы клеточного цикла
Одним из признаков, по которым можно разделить клетки, является определение фазы клеточного цикла, в которой находится клетка. Эта задача имеет практическое применение для обнаружения поврежденных клеток, которые при визуализации будут кластеризоваться отдельно от остальных. Так как клетки проходят стадии клеточного цикла непрерывно, то удобно их классифицировать, используя флуоресцентные метки. Для этого клетку окрашивают флуоресцентным красителем и используют флуоресцентный микроскоп, который фиксирует расположение и степень свечения. Сверточная сеть обучается на изображениях с этими метками и дает на выходе не только классификацию каждой клетки, а также визуализирует процесс клеточного цикла. Классификация и визуализация являются всего лишь различными способами интерпретации результатов, поэтому строятся на основе одних и тех же выведенных закономерностей[1].
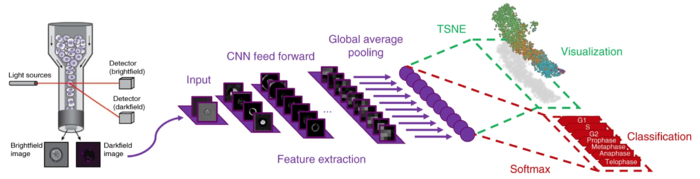
Особенностью работы данной сверточной сети является необходимость разметить только небольшую часть данных, на основании чего она далее учится размечать самостоятельно.
Идентификация раковых клеток
Другой задачей классификации клеток является обнаружение раковых клеток. Для решения этой задачи используется сверточная нейронная сеть с архитектурой VGG-16, а также трансферное обучение, то есть модель предварительно обучается на другом огромном объеме данных[2].
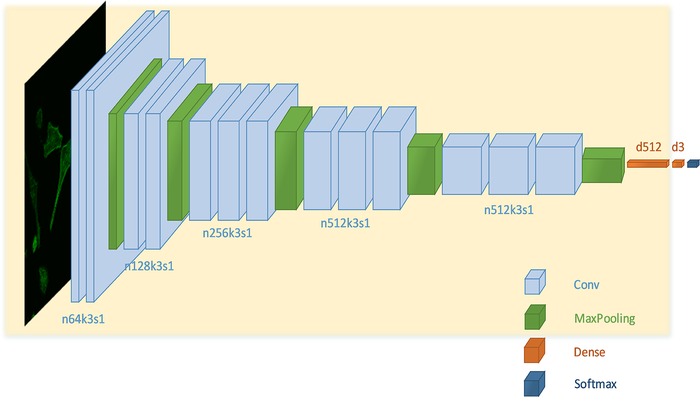
Такая сверточная сеть лучше справляется с задачей классификации клеток по сравнению с экспертом-человеком, особенно на изображениях с недостаточно хорошим качеством.
Сегментация изображений
Задача сегментации изображений, полученных с микроскопа, состоит в том, чтобы аннотировать их, то есть отмечать границы объектов (клеток, ядер). Для решения этой задачи обычно используется модифицированная полносвязная сверточная сеть U-Net[3].
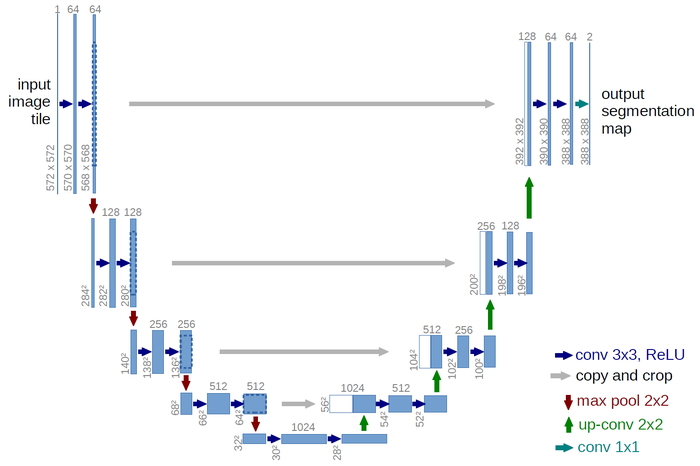
Сеть U-Net получила широкое распространение благодаря способности последовательно распознавать как большие, так и мелкие частицы, а также устойчивости к различным условиям визуализации и наборам данных.
Улучшение качества изображений
Не всегда изображения, полученные с помощью микроскопии, имеют достаточно хорошее для дальнейшей работы качество. Сверточные сети, которые улучшают качество уже имеющихся снимков, не имеют специфичных отличий.
А вот другая интересная задача направлена сразу на получение более четких изображений. Она заключается в предсказывании положения фокуса микроскопа при покадровой съемке. Для ее решения используется сверточная сеть, состоящая из двух блоков свертки и двух полносвязных блоков[4].
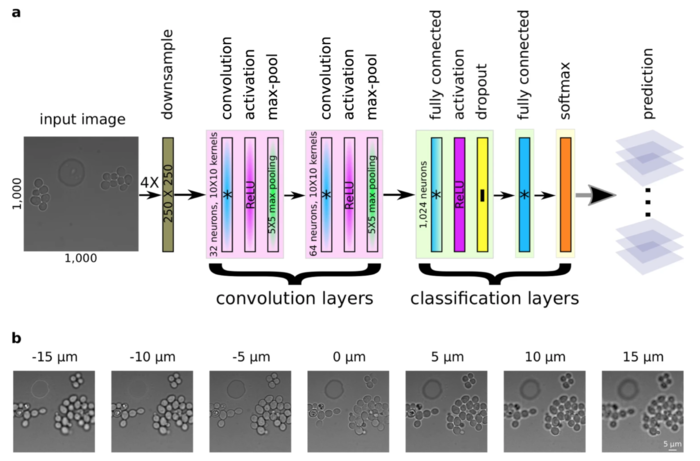
Такая сверточная сеть показывает большую точность, чем группа людей-экспертов.
См. также
Примечания
- ↑ Philipp Eulenberg — Reconstructing cell cycle and disease progression using deep learning, 2017
- ↑ Ronald Wihal Oei — Convolutional neural network for cell classification using microscope images of intracellular actin networks, 2019
- ↑ Olaf Ronneberger— Reconstructing cell cycle and disease progression using deep learning, 2015
- ↑ Ling Wei— Reconstructing cell cycle and disease progression using deep learning, 2018