Уменьшение размерности — различия между версиями
Ile86171 (обсуждение | вклад) (PCA v0.0.2) |
|||
| Строка 1: | Строка 1: | ||
| − | + | '''Метод главных компонент''' (англ. ''Principal Components Analysis, PCA'') — один из основных способов уменьшить размерность данных, потеряв наименьшее количество информации. Изобретен К. Пирсоном (англ. Karl Pearson) <ref>[https://zenodo.org/record/1430636 Pearson, K. (1901). "On Lines and Planes of Closest Fit to Systems of Points in Space"]</ref> в 1901 г. Применяется во многих областях, таких как распознавание образов, компьютерное зрение, сжатие данных и т.п. Вычисление главных компонент сводится к вычислению собственных векторов и собственных значений ковариационной матрицы исходных данных или к [[Сингулярное разложение|сингулярному разложению]] матрицы данных. Иногда метод главных компонент называют преобразованием Карунена-Лоэва (англ. ''Karhunen-Loeve'') <ref>[http://fourier.eng.hmc.edu/e161/lectures/klt/node3.html Karhunen-Loeve Transform (KLT)]</ref> или преобразованием Хотеллинга (англ. ''Hotelling transform''). | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ==Формальная постановка задачи== | |
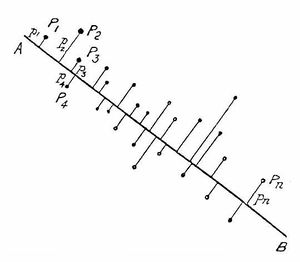
| − | + | [[File:Pearson pca example.jpg|300px|thumb|right|Иллюстрация к работе К. Пирсона (1901): даны точки <tex> P_i</tex> на плоскости, <tex> p_i</tex> — расстояние от <tex> P_i</tex> до прямой <tex> AB</tex>. Ищется прямая <tex> AB</tex>, минимизирующая сумму <tex>\sum_i p_i^2</tex>]] | |
| − | + | Пусть имеется $n$ числовых признаков $f_j(x), j = 1, ... , n$. Объекты обучающей выборки будем отождествлять с их признаковыми описаниями: $x_i \equiv (f_1(x_i), ..., f_n(x_i)), i = 1, ..., l$. Рассмотрим матрицу $F$, строки которой соответствуют признаковым описаниям обучающих объектов: | |
| + | $$F_{l \times n} = | ||
| + | \begin{pmatrix} | ||
| + | f_1(x_1) & ... & f_n(x_1)\\ | ||
| + | ... & ... & ...\\ | ||
| + | f_1(x_l) & ... & f_n(x_l) | ||
| + | \end{pmatrix} | ||
| + | = | ||
| + | \begin{pmatrix} | ||
| + | x_1\\ | ||
| + | ...\\ | ||
| + | x_l | ||
| + | \end{pmatrix}.$$ | ||
| − | + | Обозначим через $z_i = (g_1(x_i), ..., g_m(x_i))$ признаковые описания тех же объектов в новом пространстве $Z = \mathbb{R}^{m}$ меньшей размерности, $m < n$: | |
| − | |||
| − | |||
| − | + | $$G_{l \times m} = | |
| − | + | \begin{pmatrix} | |
| − | + | g_1(x_1) & ... & g_m(x_1)\\ | |
| + | ... & ... & ...\\ | ||
| + | g_1(x_l) & ... & g_m(x_l) | ||
| + | \end{pmatrix} | ||
| + | = | ||
| + | \begin{pmatrix} | ||
| + | z_1\\ | ||
| + | ...\\ | ||
| + | z_l | ||
| + | \end{pmatrix}.$$ | ||
| − | + | Потребуем, чтобы исходные признаковые описания можно было восстановить по новым описаниям с помощью некоторого линейного преобразования, определяемого матрицей $U = (u_{js})_{n \times m}$: | |
| − | |||
| − | |||
| − | |||
| − | + | $$\hat{f}_j(x) = \sum_{s = 1}^{m} g_s(x)u_{js}, \; j = 1, ..., n, \; x \in X,$$ | |
| − | |||
| − | |||
| − | + | или в векторной записи: $\hat{x} = z U^T$. Восстановленное описание $\hat{x}$ не обязано в точности совпадать с исходным описанием $x$, но их отличие на объектах обучающей выборки должно быть как можно меньше при выбранной размерности $m$. Будем искать одновременно и матрицу новых признаковых описаний $G$, и матрицу линейного преобразования $U$, при которых суммарная невязка восстановленных описаний минимальна: | |
| − | === | + | $$\Delta^2(G, U) = \sum_{i = 1}^{l} \| \hat{x}_i - x_i \|^2 = \sum_{i = 1}^{l} \| z_i U^T - x_i \|^2 = \| GU^T - F \|^2 \to \mathop{min}_{G, U},$$ |
| − | |||
| − | |||
| − | + | где все нормы евклидовы. | |
| − | + | Будем предполагать, что матрицы $G$ и $U$ невырождены: $rank \, G = rank \, U = m$. Иначе существовало бы представление $\bar{G} \bar{U}^T = G U^T$ с числом столбцов в матрице $\bar{G}$, меньшим $m$. Поэтому интересны лишь случаи, когда $m \leq rank \, F$. | |
| − | === | + | ==Решение== |
| − | |||
| − | |||
| − | + | Исчерпывающее решение сформулированной задачи даёт следующая теорема. | |
| − | + | {{Теорема | |
| + | |statement = Если $m \leq rank \, F$, то минимум $\Delta^2(G, U)$ достигается, когда столбцы матрицы $U$ есть собственные векторы $F^T F$, соответствующие $m$ максимальным собственным значениям. При этом $G = F U$, матрицы $U$ и $G$ ортогональны. | ||
| − | == | + | |proof = Запишем необходимые условия минимума: |
| − | + | ||
| − | + | <tex> | |
| − | + | \frac{\partial \Delta^2}{\partial G} = (G U^T - F) U = 0;\\ \frac{\partial \Delta^2}{\partial U} = G^T (G U^T - F) = 0. | |
| − | + | </tex> | |
| − | + | ||
| − | + | Поскольку искомые матрицы $G$ и $U$ невырождены, отсюда следует: | |
| − | + | ||
| − | + | <tex> | |
| + | G = F U (U^T U)^{-1};\\ U = F^T G (G^T G)^{-1}. | ||
| + | </tex> | ||
| + | |||
| + | Функционал $\Delta^2(G, U)$ зависит только от произведения матриц $G U^T$, поэтому решение задачи $\Delta^2(G, U) \to \mathop{min}_{G, U}$ определено с точностью до произвольного невырожденного преобразования $R: G U^T = (G R) (R^{-1} U^T)$. Распорядимся свободой выбора $R$ так, чтобы матрицы $U^T U$ и $G^T G$ оказались диагональными. Покажем, что это всегда возможно. | ||
| + | |||
| + | Пусть $\tilde{G} \tilde{U}^T$ {{---}} произвольное решение задачи. | ||
| + | |||
| + | Матрица $\tilde{U}^T \tilde{U}$ симметричная, невырожденная, положительно определенная, поэтому существует невырожденная матрица $S_{m \times m}$ такая, что $S^{-1} \tilde{U}^T \tilde{U} (S^{-1})^T = I_m$. | ||
| + | |||
| + | Матрица $S^T \tilde{G}^T \tilde{G} S$ симметричная и невырожденная, поэтому существует ортогональная матрица $T_{m \times m}$ такая, что $T^T (S^T \tilde{G}^T \tilde{G} S) T = diag(\lambda_1, ..., \lambda_m) \equiv \Lambda$ {{---}} диагональная матрица. По определению ортогональности $T^T T = I_m$. | ||
| + | |||
| + | Преобразование $R = S T$ невырождено. Положим $G = \tilde{G} R$, $U^T = R^{-1} \tilde{U}^T$. Тогда | ||
| + | |||
| + | <tex> | ||
| + | G^T G = T^T (S^T \tilde{G}^T \tilde{G} S) T = \Lambda;\\ U^T U = T^{-1} (S^{-1} \tilde{U}^T \tilde{U} (S^{-1})^T) (T^{-1})^T = (T^T T)^{-1} = I_m. | ||
| + | </tex> | ||
| + | |||
| + | В силу $G U^T = \tilde{G} \tilde{U}^T$ матрицы $G$ и $U$ являются решением задачи $\Delta^2(G, U) \to \mathop{min}_{G, U}$ и удовлетворяют необходимому условию минимума. Подставим матрицы $G$ и $U$ в | ||
| + | |||
| + | <tex> | ||
| + | G = F U (U^T U)^{-1};\\ U = F^T G (G^T G)^{-1}. | ||
| + | </tex> | ||
| + | |||
| + | Благодаря диагональности $G^T G$ и $U^T U$ соотношения существенно упростятся: | ||
| + | |||
| + | <tex> | ||
| + | G = F U;\\ U \Lambda = F^T G. | ||
| + | </tex> | ||
| + | |||
| + | Подставим первое соотношение во второе, получим $U \Lambda = F^T F U$. Это означает, что столбцы матрицы $U$ обязаны быть собственными векторами матрицы $F^T F$, а диагональные элементы $\lambda_1, ..., \lambda_m$ - соответствующими им собственными значениями. | ||
| + | |||
| + | Аналогично, подставив второе соотношение в первое, получим $G \Lambda = F F^T G$, то есть столбцы матрицы $G$ являются собственными векторами $F F^T$, соответствующими тем же самым собственным значениям. | ||
| + | |||
| + | Подставляя $G$ и $U$ в функционал $\Delta^2(G, U)$, находим: | ||
| + | |||
| + | <tex> | ||
| + | \Delta^2(G, U) = \| F - G U^T \|^2 = tr \, (F^T - U G^t)(F - G U^T) = tr \, F^T (F - G U^T) = tr \, F^T F - tr \, F^T G U^T = \| F \|^2 - tr \, U \Lambda U^T = \| F \|^2 - tr \, \Lambda = \sum_{j = 1}^{n} \lambda_j - \sum_{j = 1}^{m} \lambda_j - \sum_{j = m + 1}^{n} \lambda_j, | ||
| + | </tex> | ||
| + | |||
| + | где $\lambda_1 , ..., \lambda_n$ - все собственные значения матрицы $F^T F$. Минимум $\Delta^2$ достигается, когда $\lambda_1, ..., \lambda_m$ {{---}} наибольшие $m$ из $n$ собственных значений. | ||
| + | |||
| + | Собственные векторы $u_1, ..., u_m$, отвечающие максимальным собственным значениям, называют ''главными компонентами''. | ||
| + | }} | ||
| + | |||
| + | ==Свойства== | ||
| + | |||
| + | ===Связь с сингулярным разложением=== | ||
| + | |||
| + | Если $m = n$, то $\Delta^2(G, U) = 0$. В этом случае представление $F = G U^T$ является точным и совпадает с сингулярным разложением: $F = G U^T = V D U^T$, если положить $G = V D$ и $\Lambda = D^2$. При этом матрица $V$ ортогональна: $V^T V = I_m$. | ||
| − | + | Если $m < n$, то представление $F \approx G U^T$ является приближённым. Сингулярное разложение матрицы $G U^T$ получается из сингулярного разложения матрицы $F$ путём отбрасывания (обнуления) $n - m$ минимальных собственных значений. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ===Преобразование Карунена–Лоэва=== | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | Диагональность матрицы $G^T G = \Lambda$ означает, что новые признаки $g_1, ..., g_m$ не коррелируют на объектах из обучающей выборки. Ортогональное преобразование $U$ называют ''декоррелирующим'' или преобразованием ''Карунена–Лоэва''. Если $m = n$, то о прямое и обратное преобразование вычисляются с помощью одной и той же матрицы $U: F = G U^T$ и $G = F U$. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ===Эффективная размерность=== | |
| − | + | Главные компоненты содержат основную информацию о матрице $F$. Число главных компонент $m$ называют также ''эффективной размерностью'' задачи. На практике её определяют следующим образом. Все собственные значения матрицы $F^T F$ упорядочиваются по убыванию: $\lambda_1 \geq ... \geq \lambda_n \geq 0$. Задаётся пороговое значение $\epsilon \in [0, 1]$, достаточно близкое к нулю, и определяется наименьшее целое $m$, при котором относительная погрешность приближения матрицы $F$ не превышает $\epsilon$: | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | $$E(m) = \frac{\| G U^T - F \|^2}{\| F \|^2} = \frac{\lambda_{m + 1} + ... + \lambda_n}{\lambda_1 + ... + \lambda_n} \leq \epsilon .$$ | |
| − | |||
| − | |||
| − | |||
| − | + | Величина $E(m)$ показывает, какая доля информации теряется при замене исходных признаковых описаний длины $n$ на более короткие описания длины $m$. Метод главных компонент особенно эффективен в тех случаях, когда $E(m)$ оказывается малым уже при малых значениях $m$. Если задать число $\epsilon$ из априорных соображений не представляется возможным, прибегают к ''критерию «крутого обрыва»''. На графике $E(m)$ отмечается то значение $m$, при котором происходит резкий скачок: $E(m - 1) \gg E(m)$, при условии, что $E(m)$ уже достаточно мало. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ==Визуализация многомерных данных== | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | == | + | Метод главных компонент часто используется для представления многомерной выборки данных на двумерном графике. Для этого полагают $m = 2$ и полученные пары значений $(g_1(x_i), g_2(x_i)), i = 1, ..., l$, наносят как точки на график. Проекция на главные компоненты является наименее искаженной из всех линейных проекций многомерной выборки на какую-либо пару осей. Как правило, в осях главных компонент удаётся увидеть наиболее существенные особенности исходных данных, даже несмотря на неизбежные искажения. В частности, можно судить о наличии кластерных структур и выбросов. Две оси $g_1$ и $g_2$ отражают «две основные тенденции» в данных. Иногда их удаётся интерпретировать, если внимательно изучить, какие точки на графике являются «самыми левыми», «самыми правыми», «самыми верхними» и «самыми нижними». Этот вид анализа не позволяет делать точные количественные выводы и обычно используется |
| − | + | с целью понимания данных. Аналогичную роль играют многомерное шкалирование <ref>[https://ru.wikipedia.org/wiki/%D0%9C%D0%BD%D0%BE%D0%B3%D0%BE%D0%BC%D0%B5%D1%80%D0%BD%D0%BE%D0%B5_%D1%88%D0%BA%D0%B0%D0%BB%D0%B8%D1%80%D0%BE%D0%B2%D0%B0%D0%BD%D0%B8%D0%B5 Многомерное шкалирование]</ref> и карты Кохонена <ref>[https://ru.wikipedia.org/wiki/%D0%A1%D0%B0%D0%BC%D0%BE%D0%BE%D1%80%D0%B3%D0%B0%D0%BD%D0%B8%D0%B7%D1%83%D1%8E%D1%89%D0%B0%D1%8F%D1%81%D1%8F_%D0%BA%D0%B0%D1%80%D1%82%D0%B0_%D0%9A%D0%BE%D1%85%D0%BE%D0%BD%D0%B5%D0%BD%D0%B0 Самоорганизующаяся карта Кохонена]</ref>. | |
| − | + | ==Пример кода scikit-learn== | |
| − | < | + | Пример применения PCA к датасету Iris для уменьшения размерности: |
| − | + | <span style="color:#3D9970># Импорт библиотек</span> | |
| − | + | import numpy as np | |
| − | + | import matplotlib.pyplot as plt | |
| − | + | from sklearn import decomposition | |
| + | from sklearn import datasets | ||
| − | + | <span style="color:#3D9970># Загрузка данных</span> | |
| − | + | centers = [[1, 1], [-1, -1], [1, -1]] | |
| − | + | iris = datasets.load_iris() | |
| − | + | X = iris.data | |
| − | + | y = iris.target | |
| − | |||
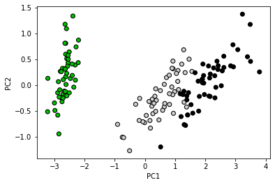
| − | < | + | [[File:Pca iris example.png|275px|thumb|right|Применения PCA к датасету Iris]] |
| − | + | <span style="color:#3D9970># Преобразование данных датасета Iris, уменьшающее размерность до 2</span> | |
| − | + | pca = decomposition.PCA(n_components=3) | |
| − | + | pca.fit(X) | |
| − | + | X = pca.transform(X) | |
| − | + | y = np.choose(y, [1, 2, 0]).astype(np.float) | |
| − | + | plt.clf() | |
| − | + | plt.cla() | |
| − | + | plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.nipy_spectral, edgecolor='k') | |
| − | + | plt.xlabel("PC1") | |
| − | + | plt.ylabel("PC2") | |
| − | + | plt.show() | |
| − | |||
==См. также== | ==См. также== | ||
| − | + | ||
| − | + | *[[Уменьшение размерности]] | |
| − | *[[ | + | *[[Сингулярное разложение]] |
| − | *[[ | + | |
| − | |||
==Примечания== | ==Примечания== | ||
<references/> | <references/> | ||
| + | |||
==Источники информации== | ==Источники информации== | ||
| − | #[http://research.cs.tamu.edu/prism/lectures/pr/ | + | |
| − | #[https://en.wikipedia.org/wiki/ | + | #[http://www.machinelearning.ru/wiki/index.php?title=%D0%9C%D0%B5%D1%82%D0%BE%D0%B4_%D0%B3%D0%BB%D0%B0%D0%B2%D0%BD%D1%8B%D1%85_%D0%BA%D0%BE%D0%BC%D0%BF%D0%BE%D0%BD%D0%B5%D0%BD%D1%82 machinelearning.ru — Метод главных компонент] |
| − | + | #[https://www.youtube.com/watch?v=wcJ0nSUr7ws Лекция "Регрессионный анализ и метод главных компонентов"] {{---}} К.В. Воронцов, курс "Машинное обучение" 2014 | |
| − | #[https://towardsdatascience.com/ | + | #[http://research.cs.tamu.edu/prism/lectures/pr/pr_l9.pdf PCA] {{---}} курс ML Texas A&M University |
| + | #[https://en.wikipedia.org/wiki/Principal_component_analysis Principal Component Analysis] {{---}} статья про Principal Component Analysis в Wikipedia | ||
| + | #[https://towardsdatascience.com/understanding-pca-fae3e243731d Understanding PCA] | ||
[[Категория: Машинное обучение]] | [[Категория: Машинное обучение]] | ||
[[Категория: Уменьшение размерности]] | [[Категория: Уменьшение размерности]] | ||
| + | [[Категория: Метод главных компонент]] | ||
Версия 02:55, 23 января 2020
Метод главных компонент (англ. Principal Components Analysis, PCA) — один из основных способов уменьшить размерность данных, потеряв наименьшее количество информации. Изобретен К. Пирсоном (англ. Karl Pearson) [1] в 1901 г. Применяется во многих областях, таких как распознавание образов, компьютерное зрение, сжатие данных и т.п. Вычисление главных компонент сводится к вычислению собственных векторов и собственных значений ковариационной матрицы исходных данных или к сингулярному разложению матрицы данных. Иногда метод главных компонент называют преобразованием Карунена-Лоэва (англ. Karhunen-Loeve) [2] или преобразованием Хотеллинга (англ. Hotelling transform).
Содержание
Формальная постановка задачи
Пусть имеется $n$ числовых признаков $f_j(x), j = 1, ... , n$. Объекты обучающей выборки будем отождествлять с их признаковыми описаниями: $x_i \equiv (f_1(x_i), ..., f_n(x_i)), i = 1, ..., l$. Рассмотрим матрицу $F$, строки которой соответствуют признаковым описаниям обучающих объектов: $$F_{l \times n} = \begin{pmatrix} f_1(x_1) & ... & f_n(x_1)\\ ... & ... & ...\\ f_1(x_l) & ... & f_n(x_l) \end{pmatrix} = \begin{pmatrix} x_1\\ ...\\ x_l \end{pmatrix}.$$
Обозначим через $z_i = (g_1(x_i), ..., g_m(x_i))$ признаковые описания тех же объектов в новом пространстве $Z = \mathbb{R}^{m}$ меньшей размерности, $m < n$:
$$G_{l \times m} = \begin{pmatrix} g_1(x_1) & ... & g_m(x_1)\\ ... & ... & ...\\ g_1(x_l) & ... & g_m(x_l) \end{pmatrix} = \begin{pmatrix} z_1\\ ...\\ z_l \end{pmatrix}.$$
Потребуем, чтобы исходные признаковые описания можно было восстановить по новым описаниям с помощью некоторого линейного преобразования, определяемого матрицей $U = (u_{js})_{n \times m}$:
$$\hat{f}_j(x) = \sum_{s = 1}^{m} g_s(x)u_{js}, \; j = 1, ..., n, \; x \in X,$$
или в векторной записи: $\hat{x} = z U^T$. Восстановленное описание $\hat{x}$ не обязано в точности совпадать с исходным описанием $x$, но их отличие на объектах обучающей выборки должно быть как можно меньше при выбранной размерности $m$. Будем искать одновременно и матрицу новых признаковых описаний $G$, и матрицу линейного преобразования $U$, при которых суммарная невязка восстановленных описаний минимальна:
$$\Delta^2(G, U) = \sum_{i = 1}^{l} \| \hat{x}_i - x_i \|^2 = \sum_{i = 1}^{l} \| z_i U^T - x_i \|^2 = \| GU^T - F \|^2 \to \mathop{min}_{G, U},$$
где все нормы евклидовы.
Будем предполагать, что матрицы $G$ и $U$ невырождены: $rank \, G = rank \, U = m$. Иначе существовало бы представление $\bar{G} \bar{U}^T = G U^T$ с числом столбцов в матрице $\bar{G}$, меньшим $m$. Поэтому интересны лишь случаи, когда $m \leq rank \, F$.
Решение
Исчерпывающее решение сформулированной задачи даёт следующая теорема.
| Теорема: |
Если $m \leq rank \, F$, то минимум $\Delta^2(G, U)$ достигается, когда столбцы матрицы $U$ есть собственные векторы $F^T F$, соответствующие $m$ максимальным собственным значениям. При этом $G = F U$, матрицы $U$ и $G$ ортогональны. |
| Доказательство: |
|
Запишем необходимые условия минимума:
Поскольку искомые матрицы $G$ и $U$ невырождены, отсюда следует:
Функционал $\Delta^2(G, U)$ зависит только от произведения матриц $G U^T$, поэтому решение задачи $\Delta^2(G, U) \to \mathop{min}_{G, U}$ определено с точностью до произвольного невырожденного преобразования $R: G U^T = (G R) (R^{-1} U^T)$. Распорядимся свободой выбора $R$ так, чтобы матрицы $U^T U$ и $G^T G$ оказались диагональными. Покажем, что это всегда возможно. Пусть $\tilde{G} \tilde{U}^T$ — произвольное решение задачи. Матрица $\tilde{U}^T \tilde{U}$ симметричная, невырожденная, положительно определенная, поэтому существует невырожденная матрица $S_{m \times m}$ такая, что $S^{-1} \tilde{U}^T \tilde{U} (S^{-1})^T = I_m$. Матрица $S^T \tilde{G}^T \tilde{G} S$ симметричная и невырожденная, поэтому существует ортогональная матрица $T_{m \times m}$ такая, что $T^T (S^T \tilde{G}^T \tilde{G} S) T = diag(\lambda_1, ..., \lambda_m) \equiv \Lambda$ — диагональная матрица. По определению ортогональности $T^T T = I_m$. Преобразование $R = S T$ невырождено. Положим $G = \tilde{G} R$, $U^T = R^{-1} \tilde{U}^T$. Тогда
В силу $G U^T = \tilde{G} \tilde{U}^T$ матрицы $G$ и $U$ являются решением задачи $\Delta^2(G, U) \to \mathop{min}_{G, U}$ и удовлетворяют необходимому условию минимума. Подставим матрицы $G$ и $U$ в
Благодаря диагональности $G^T G$ и $U^T U$ соотношения существенно упростятся:
Подставим первое соотношение во второе, получим $U \Lambda = F^T F U$. Это означает, что столбцы матрицы $U$ обязаны быть собственными векторами матрицы $F^T F$, а диагональные элементы $\lambda_1, ..., \lambda_m$ - соответствующими им собственными значениями. Аналогично, подставив второе соотношение в первое, получим $G \Lambda = F F^T G$, то есть столбцы матрицы $G$ являются собственными векторами $F F^T$, соответствующими тем же самым собственным значениям. Подставляя $G$ и $U$ в функционал $\Delta^2(G, U)$, находим:
где $\lambda_1 , ..., \lambda_n$ - все собственные значения матрицы $F^T F$. Минимум $\Delta^2$ достигается, когда $\lambda_1, ..., \lambda_m$ — наибольшие $m$ из $n$ собственных значений. Собственные векторы $u_1, ..., u_m$, отвечающие максимальным собственным значениям, называют главными компонентами. |
Свойства
Связь с сингулярным разложением
Если $m = n$, то $\Delta^2(G, U) = 0$. В этом случае представление $F = G U^T$ является точным и совпадает с сингулярным разложением: $F = G U^T = V D U^T$, если положить $G = V D$ и $\Lambda = D^2$. При этом матрица $V$ ортогональна: $V^T V = I_m$.
Если $m < n$, то представление $F \approx G U^T$ является приближённым. Сингулярное разложение матрицы $G U^T$ получается из сингулярного разложения матрицы $F$ путём отбрасывания (обнуления) $n - m$ минимальных собственных значений.
Преобразование Карунена–Лоэва
Диагональность матрицы $G^T G = \Lambda$ означает, что новые признаки $g_1, ..., g_m$ не коррелируют на объектах из обучающей выборки. Ортогональное преобразование $U$ называют декоррелирующим или преобразованием Карунена–Лоэва. Если $m = n$, то о прямое и обратное преобразование вычисляются с помощью одной и той же матрицы $U: F = G U^T$ и $G = F U$.
Эффективная размерность
Главные компоненты содержат основную информацию о матрице $F$. Число главных компонент $m$ называют также эффективной размерностью задачи. На практике её определяют следующим образом. Все собственные значения матрицы $F^T F$ упорядочиваются по убыванию: $\lambda_1 \geq ... \geq \lambda_n \geq 0$. Задаётся пороговое значение $\epsilon \in [0, 1]$, достаточно близкое к нулю, и определяется наименьшее целое $m$, при котором относительная погрешность приближения матрицы $F$ не превышает $\epsilon$:
$$E(m) = \frac{\| G U^T - F \|^2}{\| F \|^2} = \frac{\lambda_{m + 1} + ... + \lambda_n}{\lambda_1 + ... + \lambda_n} \leq \epsilon .$$
Величина $E(m)$ показывает, какая доля информации теряется при замене исходных признаковых описаний длины $n$ на более короткие описания длины $m$. Метод главных компонент особенно эффективен в тех случаях, когда $E(m)$ оказывается малым уже при малых значениях $m$. Если задать число $\epsilon$ из априорных соображений не представляется возможным, прибегают к критерию «крутого обрыва». На графике $E(m)$ отмечается то значение $m$, при котором происходит резкий скачок: $E(m - 1) \gg E(m)$, при условии, что $E(m)$ уже достаточно мало.
Визуализация многомерных данных
Метод главных компонент часто используется для представления многомерной выборки данных на двумерном графике. Для этого полагают $m = 2$ и полученные пары значений $(g_1(x_i), g_2(x_i)), i = 1, ..., l$, наносят как точки на график. Проекция на главные компоненты является наименее искаженной из всех линейных проекций многомерной выборки на какую-либо пару осей. Как правило, в осях главных компонент удаётся увидеть наиболее существенные особенности исходных данных, даже несмотря на неизбежные искажения. В частности, можно судить о наличии кластерных структур и выбросов. Две оси $g_1$ и $g_2$ отражают «две основные тенденции» в данных. Иногда их удаётся интерпретировать, если внимательно изучить, какие точки на графике являются «самыми левыми», «самыми правыми», «самыми верхними» и «самыми нижними». Этот вид анализа не позволяет делать точные количественные выводы и обычно используется с целью понимания данных. Аналогичную роль играют многомерное шкалирование [3] и карты Кохонена [4].
Пример кода scikit-learn
Пример применения PCA к датасету Iris для уменьшения размерности:
# Импорт библиотек
import numpy as np
import matplotlib.pyplot as plt
from sklearn import decomposition
from sklearn import datasets
# Загрузка данных
centers = [[1, 1], [-1, -1], [1, -1]]
iris = datasets.load_iris()
X = iris.data
y = iris.target
# Преобразование данных датасета Iris, уменьшающее размерность до 2
pca = decomposition.PCA(n_components=3)
pca.fit(X)
X = pca.transform(X)
y = np.choose(y, [1, 2, 0]).astype(np.float)
plt.clf()
plt.cla()
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.nipy_spectral, edgecolor='k')
plt.xlabel("PC1")
plt.ylabel("PC2")
plt.show()
См. также
Примечания
Источники информации
- machinelearning.ru — Метод главных компонент
- Лекция "Регрессионный анализ и метод главных компонентов" — К.В. Воронцов, курс "Машинное обучение" 2014
- PCA — курс ML Texas A&M University
- Principal Component Analysis — статья про Principal Component Analysis в Wikipedia
- Understanding PCA